概要
海洋および淡水環境(太平洋、東シナ海、東京湾、荒川)における放線菌群集構造を、文化に依存しない分子法によって調査し、空間的および季節的分布を明らかにしました。DNAフラグメントを増幅し、変性剤濃度勾配ゲル電気泳動(DGGE)および多次元尺度構成法(MDS)分析とそれに続くシーケンシング分析によって分析されました。
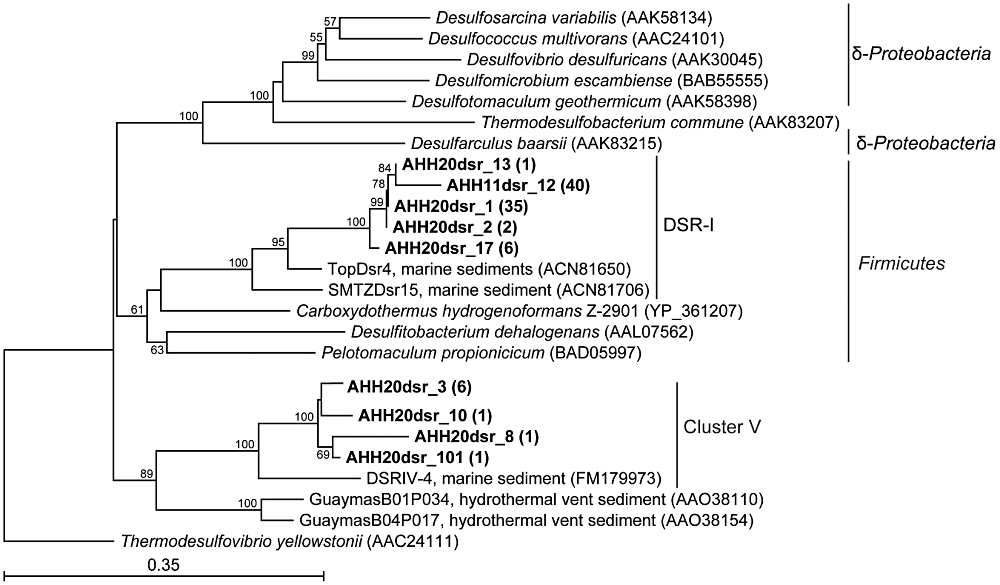
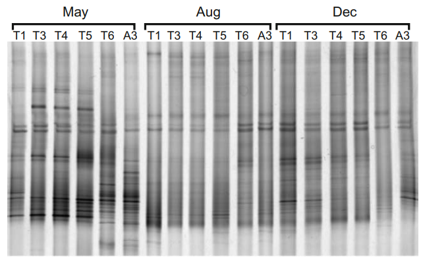
放線菌群集の構造は、太平洋、東シナ海、東京湾、荒川の各観測所で異なり、各地域で異なる個体群が再建されました。太平洋と東シナ海の表面と深さ100mの間で放線菌群集に垂直方向の変動があったが、群集は深さ200mから1,000mまで類似していました。東京湾のコミュニティにも明確な季節変動がありました。 DGGEバンドから回収されたDNAフラグメントの系統発生分析により、主要な放線菌株のほとんどは培養されておらず、Streptomyces、Micromonospora、Microbispora、Salinispora、Actinoplanesグループなどのよく知られた培養可能な株とはかなり異なることが明らかになりました。以上の結果から、海洋環境が生物活性化合物を生成する新しい放線菌集団を発見するための魅力的なターゲットであり、サンプリングの深さと季節がさまざまな集団を効果的に隔離するための重要な考慮事項であることが示唆されました。
雑誌名
Marine Biotechnology
論文タイトル
Actinomycetal community structures in seawater and freshwater examined by DGGE analysis of 16S rRNA gene fragments
著者
Akihiro Yoshida, Yuna Seo, Shuhei Suzuki, Tomohiko Nishino, Takeshi Kobayashi, Naoko Hamada-Sato, Kazuhiro Kogure, Chiaki Imada
DOI
引用
Yoshida, Akihiro, Yuna Seo, Shuhei Suzuki, Tomohiko Nishino, Takeshi Kobayashi, Naoko Hamada-Sato, Kazuhiro Kogure, and Chiaki Imada. “Actinomycetal community structures in seawater and freshwater examined by DGGE analysis of 16S rRNA gene fragments.” Marine Biotechnology 10, no. 5 (2008): 554-563.